CRISPR
CRISPR ( C lustered R egularly I nterspaced S Hort P alindromic R epeats) olyan részei a repetitív DNS ( ismétlődések ), amely a genom számos baktérium és archaea fordulnak elő. Olyan mechanizmust szolgálnak, a CRISPR / Cas rendszert, amely ellenáll az idegen gének vírusok vagy plazmidok általi behatolásának, ezért számos prokarióta immunrendszerének megfelelő része . Ez a rendszer képezi a géntechnológiai CRISPR / Cas módszer alapját a géntechnológiával módosított szervezetek előállításához .
Felfedezés és tulajdonságok
Ismétlődő DNS szakaszok létezését, amelyet ma CRISPR néven ismerünk, 1987-ben fedezték fel Yoshizumi Ishino és munkatársai az Escherichia coli K12 baktérium törzsben . 29 nukleotid ismétlődő szekvenciáját azonosították , amelyeket 32 nukleotid variábilis régiói szakítottak meg. 1993-ban hasonló régiókat fedeztek fel a Mycobacterium tuberculosis DNS-en, és "közvetlen változó ismétléseknek" (DVR) nevezték őket. 1995-ben ezeket a szekvenciákat a Haloferax volcanii és a Haloferax mediterranei tengeri baktériumokban is felfedezte Francisco Mojica spanyol mikrobiológus , aki "Tandem ismétlések" (TREP). A Mojica munkacsoport más baktériumokat és archeákat azonosított a megfelelő szekvenciákkal, és új nevet választott ezeknek az azonos ismétléseknek, mint „Short Regularly Spaced Repeats” (SRSR). A szakirodalomban további neveket adtak hozzá, amelyek szintén hivatkoztak ezekre a szekvenciákra, például "távtartó közbeiktatott és közvetlen ismétlések" (SPIDR) és "hosszú fürtözött tandem ismétlések" (LCTR). Jansen és munkatársai 2002-ben használták először a „Clusterered Regularly Interspace Short Palindromic Repeats” (röviden CRISPR) kifejezést. Ismertté vált, hogy sokféle prokarióta genomjában hasonló struktúrák léteznek, és felfedeztek egy olyan géncsoportot, amely az összes vizsgált organizmusban közel állt a CRISPR gén lokuszához , ezért cas géneknek nevezték őket (CRISPR-asszociáltak). Jansen és munkatársai négy különböző Cas mag szekvenciát azonosítottak (Cas1 – Cas4), 2005-ig összesen 41 megfelelő gént és további két Cas mag szekvenciát (Cas5 és Cas6), valamint a CRISPR / Cas-Systems összesen nyolc altípusát ismertették.
Ma már ismert, hogy az eddig szekvenált baktériumok körülbelül 45% -ának és az archeák 83% -ának genomja legalább egy CRISPR szerkezetet tartalmaz.
A Francisella faj kórokozói a CRISPR-Cas rendszert használják az immun megkerülésére . A Neisseria meningitidis és a Campylobacter jejuni esetében a rendszer egy korábban ismeretlen mechanizmusú patogenitási tényező .
szerkezet
A CRISPR lókusz lényegében két fő összetevőből áll: a CAS gének tartalmazó cas - Operon és a CRISPR tömb, amely áll egy leader tagjai (más néven repeat-spacer-array) szekvenciát, és egy ismételt távtartó szekvenciát.
Ismételje meg a távtartó szekvenciát
Az ismétlődő alapmotívum ( ismétlések ) egyes szekvenciáinak hossza 23 és 47 bp között változik. Az ismétlések váltakoznak a távtartókkal , amelyek hossza 21–72 bp. Míg az ismétlődő szekvencia megmarad egy CRISPR struktúrán belül, a CRISPR szekvenciája a különböző mikroorganizmusokban nagyon eltérő. A baktériumok CRISPR ismétléseinek szekvenciája általában palindromikus (azaz tükörbe fordított komplementer), ami a társított RNS stabil másodlagos szerkezetét eredményezi , míg az archeák legtöbb ismétlődése nem palindrom.
A távtartó szakaszok szekvenciája széles körben változik, mind a CRISPR struktúrán belül, mind a különböző prokariótákban . 2005-ben kiderült, hogy a távtartó szekvenciák megegyeznek a bakteriofágokból és plazmidokból származó idegen DNS- ekkel. Ez ahhoz a hipotézishez vezetett, hogy a CRISPR feladata a szervezet védelme az idegen DNS ellen.
cas operon
A cas operon szintén része a CRISPR gén lokuszának . A cas operon olyan cas géneket és kódolandó fehérjéket tartalmaz , amelyek szükségesek az adaptív immunválaszhoz, pl. B. helikázok , nukleázok , de az RNS-kötődésre jellemző tulajdonságú fehérjék is. A cas -Gene két modulra osztható: az effektorra és az adaptációs modulra . Az effektor modul a cas gének egy csoportja, amelyet a genetikai anyag azonosítására használnak . Az adaptációs modul cas géneket is tartalmaz , és az effektor fehérjék segítségével hozzájárul a baktériumok genomjába integrálható protospacerek kiválasztásához.
vezető sorrend
Az ismétlődő spacer szekvencia közelében van egy úgynevezett vezető szekvencia (nem tévesztendő össze az mRNS vezető szekvenciájával ). A vezető szekvencia egy adenin és thymine- gazdag szekvencia egy 100-500 bp hosszúságú. Az ismétlésekhez hasonlóan a vezérszekvenciák is körülbelül 80% -ban azonosak egy genomban, de jelentős különbségeket mutatnak a különböző organizmusokon belül. A nem-kódoló szekvenciát, úgy lehet két területre osztható: egy mag vezetője és egy kiterjesztett vezetője . A mag vezető őrzi számos organizmusból, és hosszuk 20-300 bp általában rövidebb, mint a kiterjesztett vezetője . Ezenkívül a magvezető rendelkezik egy promoterelemmel , amelyhez a szabályozó fehérjék kötődhetnek annak érdekében, hogy képesek legyenek szabályozni a génexpressziót , pontosabban a CRISPR transzkripció iniciálását és a spacer megszerzését.
Az a kiterjesztett vezető , amelynek hossza 50-500 bp-mal hosszabb, mint a mag vezetője, és a CRISPR-től távoli, konzervált szekvenciákat is tartalmaz, feltehetően gén duplikációval . A kibővített vezető funkciói jelenleg ismeretlenek. A kibővített vezetőnek feltehetően nincs fontos funkciója.
Immunitás a CRISPR-en keresztül
A CRISPR-en keresztüli immunitás három lépésben megy végbe, ahol az utolsó két lépés különbözik az adott CRISPR / Cas rendszertípusoktól.
1. Alkalmazkodás: Barrangou és munkatársai 2007-ben kimutatták, hogy a fágokkal fertőzött baktériumok az idegen DNS részeit távtartóként integrálhatják genomjuk CRISPR régióiban, és ezáltal immunitást alakíthatnak ki a fágokkal szemben . Megmutatták azt is, hogy a baktériumok CRISPR régióiba mesterségesen beillesztett távtartó szekvenciák ellenállóvá teszik őket a kapcsolódó fágokkal szemben. Ha a távtartó szekvenciákat ismét kivágják, az ellenállás is megszűnik. Kimutatták azt is, hogy a cas gének alapvető szerepet játszanak a fágvédelemben: egyes cas gének (cas1) inaktiválása megakadályozza a fágok védelmét a távtartók jelenléte ellenére. Más cas gének (cas7) aktivitása szükséges az új távtartók integrálásához a CRISPR szekvenciába.
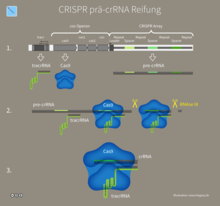
2. crRNS transzkripció és feldolgozás: A CRISPR gén lokuszát átírják pre-crRNS-be, majd érett crRNS-vé dolgozzák fel.
3. Interferencia: Az érett crRNS egy Cas fehérjéhez vagy egy Cas fehérje komplexhez kapcsolódik, és ez interferencia komplex kialakulásához vezet. A CRISPR / Cas rendszer I és II típusú, kölcsönhatása az interferencia komplex a PAM szekvencia motívum az a fág DNS-t vezet a lebomlását a DNS-t a segítségével a Cas3 az I. típusú és a Cas9 a II típusú, míg a III típusú nincs szükség PAM-ra, és az RNS is lebontható a DNS mellett.
A koevolúció során a bakteriofágok anti-CRISPR fehérjéket fejlesztettek ki az immunrendszer gátlására.
gépezet
Az elmúlt évek nagy előrelépése ellenére még nem teljesen ismert a mechanizmus, amely révén a CRISPR / Cas rendszer immunitást nyújt a prokarióták ellen. Feltételezzük, hogy az immunizálási folyamatban az exogén DNS-t egy Cas fehérje komplex felismeri, és új távtartóként integrálja a CRISPR területekre. Hogy ezek a folyamatok hogyan működnek részletesen, még nem teljesen tisztázott.
alkalmazkodás
A CRISPR / Cas rendszerek képesek módosítani a baktériumok és az archeák genomját azáltal, hogy idegen DNS-szekvenciákat, úgynevezett távtartókat integrálnak a CRISPR-tömb ismétlődései közé. Ezt a folyamatot adaptációnak vagy távtartó szerzésnek nevezik. Az adaptáció két szakaszra osztható:
- Idegen DNS távtartó szekvenciáinak (úgynevezett protospacerek) rögzítése,
- Spacer integráció.
Az adaptáció mechanizmusát - néhány kivételtől eltekintve - részletesen megvizsgálták az I. típusú E. coli CRISPR / Cas rendszerben (más néven CRISPR / Cas rendszerű IE típusban). Az adaptáció fő szereplőit a cas1 és a cas2 gének kódolják , amelyek konzerválódnak a különböző CRISPR / Cas rendszertípusokban.
Az adaptáció első fázisa, a távtartó szekvenciák befogása az idegen DNS-ből kétféle módon történhet az I. típusú CRISPR / Cas rendszerrel: naiv vagy alapozott . A naiv adaptációban csak a Cas1 és a Cas2 fehérjékre van szükség a távtartók elfogadásához előítéletek nélkül, míg a primer adaptáció a meglévő távtartóktól ( primer távtartóktól ) függ, és előzetes szelekciót végeznek arról, hogy mely távtartók vannak beépítve a genomba. Ehhez a Cas1 és Cas2 fehérjék mellett egy Cas fehérjékből (I. típusú interferencia komplex, Cascade ) és a Cas3 nukleázból álló fehérjekomplex is szükséges. A CRISPR / Cas rendszerek más típusai további fehérjéket kódolnak az adaptációhoz.
A primer adaptáció mechanizmusa a crRNS-hez kötött Cascade fehérjekomplexum ( CRISPR-asszociált komplex vírusellenes védekezéshez ) kötésével kezdődik a betolakodó DNS Protospacer Adjacent Motifjéhez (PAM) a megkönnyített 1D diffúzió kombinációja révén (végigcsúszva) a DNS) és a 3D diffúzió ( ugrálás ). Miután a DNS-t meghajlította és lecsavarta a Cascade, egy R-hurok képződik a crRNS és a Cascade-hez kötött DNS-szál komplementer bázispárosításával . Amikor a Protospacert a Cascade teljesen letekeri, az R-hurok teljesen kialakul. Az R-hurok teljes kialakulása a Cascade konformációjának megváltozásához vezet, és ezáltal a Cas3-nak a Cascade-hoz való kötődését okozza. Ezenkívül az R hurok teljes kialakulása kidudorodást vált ki a kötetlen szálon, és ezáltal lehetővé teszi a Cas3 átvágását ezen a dudoron. A Cas3 által generált egyszálú fragmenseket ezután a Cas1-Cas2 komplex egyszálú protospacerekké dolgozza fel. Az egyszálú protospacerré történő feldolgozás után egy teljes vagy részben kettős szálú protospacerré alakul, így lehetséges a CRISPR tömbbe történő integrálás. A CRISPR-en keresztüli immunitás utolsó lépése után is lehetséges az interferencia, a távtartó szekvenciák befogása. A lebomlott DNS fragmenseit a RecBCD enzim vagy más nukleázok révén protospacerekké alakítják, és a Cas1-Cas2 komplex segítségével integrálják a CRISPR tömbbe (naiv adaptáció).
A távtartó integrációja nem önkényesen történik a CRISPR tömbben, hanem polarizált, azaz Ez azt jelenti, hogy a távtartók a CRISPR tömb egy meghatározott pontjában vannak, pontosabban a vezető szekvencia közelében. Ez a mechanizmus biztosítja, hogy az új távtartók mindig integrálódjanak a vezetőszekvencia közelébe, és a távtartók időrendi integrációja optimalizálja az adaptív immunválaszt a legújabb vírusfertőzésekre. A CRISPR / Cas rendszer I típusú igényli a fehérje integrációs gazdafaktor (IHF), amely kötődik a vezető szekvenciát. Ennek eredményeként a vezető szekvencia körülbelül 120 ° -kal meghajlik, és létrehoz egy kötési helyet a Cas1-Cas2 komplex számára, így a komplex a vezérszekvenciához legközelebb eső ismétlés közelében helyezkedik el . Ennek eredményeként a vezető ismétlés határa a távtartó integrációjának helyévé válik. A II. Típusú CRISPR / Cas rendszerrel a távtartó integráció is polarizált, de további fehérjék használata nélkül. A II. Típusú Cas1-Cas2 komplex Cas1 α- spirálja a vezető szekvencia kicsi hornyához kötődik , amely más néven vezető-lehorgonyzó szekvencia (LAS). A Cas1 LAS-kölcsönhatásban lévő tartományának rugalmassága miatt a távtartó integrációnak nem feltétlenül a vezető- ismétlés határán kell végbemennie, hanem egy távtartó-ismétlés határán is megvalósulhat. Mutált LAS esetén ez méhen kívüli távtartó integrációhoz vezethet, a távtartókat pedig a CRISPR tömb közepére integrálva.
A E. coli , a távtartó integrációs keresztül zajlik két transesterifications , az első átészterezés megy végbe, a nukleofil támadását a hidroxilcsoport a 3 „végén az egyik szálával protospacer a vezetőpeptid ismétlési határ és ezáltal képződéséhez vezet egy fél helyszínről - az integrációt közvetítő vezet. Az első átészterezés az ismétlés inflexióját hozza létre, amely lehetővé teszi a második átészterezést. Az átmenet a teljesen integrált távtartóba, a teljes helyű termékbe egy második átészterezés útján történik, a hidroxilcsoport nukleofil támadása a prototérköz ellentétes szálának 3'-végén az ismétlődő-távtartó határ közelében történik. A második átészterezést egy úgynevezett vonalzó mechanizmus szabályozza. Az E. coliban az ismétlés két inverz ismétlést (IR) tartalmaz, amelyek kódolják a szerkezeti motívumokat, és lehorgonyzóként szolgálnak az úgynevezett „molekuláris vonalzók” számára. Ezek a molekuláris uralkodók biztosítják, hogy a második nukleofil támadás csak az ismétlés-távtartó határ közelében következik be, és hogy az ismétlés hossza megmaradjon a távtartó integrálása és az ismétlés duplikálása után. Az átészterezés után keletkező DNS-réseket különféle DNS-javító mechanizmusok zárják le , ideértve a homológiával irányított javítást (HDR), a nem homológ végcsatlakozást (NHEJ) és a mikrohomológia által közvetített végcsatlakozást (MMEJ). A távtartó integrálása után a vezérszekvenciával szomszédos ismétlést azonos hosszúsággal másoltuk.
crRNS transzkripció és feldolgozás
Az érett CRISPR-RNS (crRNS) biogenezise három lépésben mehet végbe, és részben egyedi távtartó szekvenciájának segítségével egy vagy több Cas fehérjét vezet a behatoló nukleinsavhoz , amelyet a genetikai anyag lehetséges lebontására használnak. szekvencia-specifikus RNS felismerés után.
- Hosszú elsődleges transzkriptum, a prekurzor crRNS (pre-crRNS) átírása a vezető szekvenciában található promóterrel .
- Az pre-crRNS elsődleges hasítása meghatározott helyeken, egy teljes távtartó szekvenciával rendelkező részleges ismétlődő szekvenciákkal rendelkező crRNS előállításához.
- Bizonyos esetekben további szekunder hasításra van szükség az aktív érett krRNS előállításához.
Az I. és a III. CRISPR / Cas rendszerben a Cas6 család specifikus endoribonukleázára van szükség, vagy alternatív megoldásként az Cas5d IC típusra, amely önmagában vagy más Cas fehérjékkel komplexben hasítva hasítja az pre-crRNS-t az ismétlődő régiókban. A II. Típusban a tracrRNS a pre-crRNS hasítását az ismétlődő régiókban a III endoribonukleáz ( RNáz III ) segítségével Cas9 jelenlétében transzaktiválja.
Amikor az I. típusú CRISPR / Cas rendszer, az pre-crRNS endoribonukleotiddal történő feldolgozása felszabadítja a fémfüggetlen Cas6 családot (vagy alternatívaként az IC típusú Cas5d-vel), amely a megismételt szekvenciát konzervált pozíciókban, általában 8 nt- rel felfelé ("upstream"), az 5'-vég irányában) az ismétlődő-távtartó határtól elszakad. Míg az IC, IE és IF típusokhoz csak egy érlelési lépés szükséges, addig az IA, IB és ID típusokhoz egy második érlelési lépés szükséges, amelyek összetevői és mechanizmusa jelenleg ismeretlen. Az IC, ID, IE és IF típusú pre-crRNS palindrom ismétlődéseinek hajtűszerkezete van, amelyek feltárják a hasítási helyeket a megfelelő endoribonukleáz katalitikus doménjéhez. A hasítás után a hajtűszerkezetek továbbra is társulnak a megfelelő endoribonukleázzal, és a Cascade alegységei kötődnek az 5'-végén lévő szekvenciához és a távtartóhoz, amelyeket a genetikai anyag felismerésére használnak.
A CRISPR / Cas rendszer II típusú, miután transzkripcióját CRISPR tömb és a tracrRNA, bázis párosodás az anti-ismétlődését tracrRNA az ismétlési a pre-crRNA és a kialakulását a tracrRNA: pre-crRNA duplex, amely a Cas9 által stabilizált, zajlik. Ezenkívül a duplex képződés az RNáz III toborzásához és így a duplex együttes feldolgozásához vezet. Ezt követi a második érlelési lépés exonukleázzal történő vágással és / vagy endoribonukleázzal történő hasítással. A II-C típus egy érett krRNS alternatív szintézisútját jelenti, a promóterek a CRISPR tömb ismétlődésein belül helyezkednek el, és egy rövid pre-crRNS transzkriptum képezhető úgy, hogy az RNáz III által történő hasítás már nem szükséges. Az érett duplex a Cas9-gyel komplexálódik, és egy II. Típusú interferencia-komplexet alkot, amely felismeri és hasítja a kettős szálú DNS-t (dsDNS).
A III. Típusú CRISPR / Cas rendszerben az pre-crRNS hasítása az ismétlésekben a Cas6 által történik, és ezáltal olyan crRNS közbenső termékeket hoz létre, amelyek mindegyikének részleges szekvenciája van az pre-crRNS ismétlődéseinek 5 'és 3' végén. (1X közbenső). Ezt követően a III-A-ban az 1X köztitermék komplexet képez a Csm komplextel és III-B csoportban a Cmr komplexel. Ezután a második érlelési lépést úgy végezzük , hogy a 3'-végén levágjuk a még nem azonosított nukleázokat az érett crRNS kialakításához.
interferencia
Az integrált vírus távtartó szekvenciákat tartalmazó pre-crRNS érett krRNS-vé történő feldolgozása után egy CRISPR ribonukleoprotein komplexhez (crRNP) társul és interferencia komplexet (más néven CRISPR felügyeleti komplexet) képez egy újabb fertőzés után, a vírus DNS vagy RNS szekvencia-specifikus módon lebontható. Az interferencia mechanizmusát az összes CRISPR / Cas rendszertípusban bizonyos kulcsfontosságú fehérjék jellemzik: Cas 3 (I. típus), Cas 9 (II. Típus) és Cas10 (III. Típus), és főleg a crRNP komplex felépítésében különböznek (crRNP összeállítás ) valamint a genetikai anyag lebontásának mechanizmusában. Az összes I. típusú crRNP-komplexet Cascade-nak nevezik, míg a II-es típusban a Cas9 fehérje, mint egyetlen fehérje felelős a nukleinsav hasításáért. A III. Típusban a Csm (III-A típus) és a Cmr (III-B típus) crRNP komplexek felelősek az interferenciáért.
Az I. típusú CRISPR / Cas rendszer esetén az interferencia öt lépésben történik:
- Cascade szerelés
- PAM kimutatás és megtartás
- R-hurok kialakulása
- Cas3 toborzás
- DNS lebontása
Az előzetes crRNS feldolgozása után az érett E. coli- ból származó crRNS egy 5 ' nyélből (8 nt), egy hidroxilcsoportból, egy távtartó szekvenciából (32 nt) és egy hajtű szerkezetből áll a 3' végén (21 nt) a 2'-3'-ciklikus foszfátvég, a Cas6e feldolgozás után a hajtűszerkezettel társul. Az érett crRNS hasítása után megtörténik a kaszkád összeállítás, az első lépés a végződés lezárása . A Cas5 az 5'- fogantyúhoz kötődik, és így kezdetben horogszerű struktúrát hoz létre a crRNS-ből. Ezenkívül a Cas7 fehérje hat példánya kötődik a távtartó szekvenciához, így az úgynevezett Cas7 gerincet kapja . Különleges, hogy a Cas5 és a Cas7 szerkezete úgynevezett konzervált „tenyér-hüvelykujj doménnel” rendelkezik, amelyek hozzájárulnak a Cas7 gerincének átszövéséhez. A Cas5e vagy mind a hat Cas7 alegység (Cas7.1 - Cas7.6) „hüvelykujja” (β-hajtűszerkezet) egy bizonyos helyzetben és 6 nt intervallumon belül összekapcsolja az 5'- nyél crRNS-jét. távtartó szekvenciát, és biztosítja, hogy az összekapcsolt nukleotidok deformált konfigurációt kapjanak, és már nem alkalmasak a bázis és a cél DNS párosítására. Ezzel szemben a szomszédos 5-nt szekvenciák minden törésnél kinyúlnak, és megtartják szakaszos A-DNS- szerű alakjukat, így ezek a szekvenciák alkalmasak a bázis és a cél-DNS párosítására. Ezután két további fehérje, a Cse1 (nagy alegység) és a Cse2 dimer (kis alegységek) kötődik a Cas7 alegységekhez fehérje-fehérje kölcsönhatás révén . Mindkét fehérje részt vesz a DNS-kötésben, a nagy alegység szintén hozzájárul a célszelekcióhoz. Ez biztosítja, hogy az interferencia komplex mindig keresse a sejtet a potenciális cél DNS után. A végső összeszerelés után a Cascade-t gyakran csikóhal- szerű szerkezetként írják le .
Most a Cascade segítségével a cél DNS keresése zajlik, a Pse azonosításáért a Cse1 L1 hurka felel. Az IE típusban a PAM azonosítása után a kettős szálú vírus DNS bejut a Cas7.5 és Cas7.6 közötti résbe, majd továbbjut a nagy alegységre (Cse1), amely azonban főleg nem specifikus kölcsönhatásban áll a cél DNS. A PAM felismerés által L1 hurok Cse1 destabilizálja a kettős-szálú DNS-t, úgy, hogy a bázispárosodás közötti 7 nt hosszú vetőmag régió a PAM-határos DNS protospacer szekvenciát a crRNA kerülhet sor. Az ezt követő R-hurok kialakulása a crRNS-távtartó teljes bázispárosításával a vírusprotoszférával ugyanazon mechanizmus szerint megy végbe, mint a távtartószekvenciák rögzítésekor. A teljes R-hurok kialakulása után a nagy és a kis alegység konformációja megváltozik, így interakciós helyek jönnek létre a nagy alegységen a Cas3 C- terminális doménjához (CTD). A Cas3 toborzása a kettéágazásnál a CTD disszociálásával megnyitja a csatornát a kétszálú DNS számára . Miután a dsDNS felgyülemlett a csatornában, a csatornát a CTD újrapozícionálásával lezárjuk, és a kettős szálú DNS nem kötött szálát a Cas3 HD nukleáz doménjében tároljuk, ahol a vágás megtörténik. A vágást a PAM-tól lefelé körülbelül 11–15 nm- re végezzük két katalitikus átmenetifém- ion felhasználásával. A Cas3 konformációs változása a helika részen, amelyet a vágás vált ki (a RecA-szerű doménből ( RecA ) és a RecA-szerű doménból 2 ( RecA2 ) áll) az ATP-t megköti és hidrolizálja , a dsDNS letekercseléséhez felszabaduló energia 3 ′ → 5 ′ irányt használunk. A letekerés a RecA2 hajtűszerkezetén történik. A helica rész mozgatásával ez kiváltja a HD domén elmozdulását új szubsztrátokba a további exonukleolitikus lebontás érdekében. A lebontás után képződött egyszálú DNS-t (ssDNS) a Cas3 szintén exonukleolitikusan lebontja. Így a megcélzott DNS hatékonyan eltávolítható a sejtből, és a Cascade újrahasznosítható a további PAM-felismerés érdekében.
A CRISPR / Cas II típusú rendszer esetén az interferencia négy lépésben történik:
- Az aktív II típusú CRISPR felügyeleti komplex kialakulása
- PAM kimutatás és megtartás
- R-hurok kialakulása
- DNS lebontása
Három független tanulmány a S. Pyogenes Cas9 szerkezetéről azt mutatja, hogy a Cas9 két lebenyből áll, amelyek együttesen egy félhold alakot fogadnak el. A REC lebeny ( felismerési lebeny ) egy hosszú α-hélixből (híd spirál), egy Rec2 doménből és egy Rec1 doménből áll a tracrRNS: crRNS duplex felismerésére. A NUC lebeny (angol nukleázlebeny ) két nukleázdoménből áll a DNS hasításhoz, amelyeket HNH-nak (jellegzetes hisztidin- és aszparaginmaradványokról neveznek el) és RuvC-nek ( a DNS-hez kapcsolt E. coli fehérje után neveznek el . ismert, és egy további C- terminális topoizomeráz - homológiai domén (CTD), amely a PAM felismerés megkönnyítéséhez szükséges.
A Cas9 aktiválása a duplex Rec1-hez való kötésével, amely az interferencia előtt történt, a HNH konformációs változását indította el, ami a REC lebeny helyzetének megváltozásához és egy központi pozitív töltésű csatorna kialakulásához vezetett a behatoló DNS számára. . A duplex együttes feldolgozása után létrejött aktív II típusú CRISPR megfigyelő komplex kész PAM szekvenciával rendelkező vírus DNS keresésére. A PAM-kötés után a DNS lokálisan megolvad . Itt nem párosulnak nukleáris bázisok , úgynevezett olvadt buborékok (angolul. Megolvadt buborékok ), amelyek az R-hurok képződéséig képződnek egy PAM proximális, 8-12 hosszú mag hozzájárul a DNS szekvenciájához. Ezután mindegyik nukleázdomén hasított egy DNS-szálat Mg 2+ ionok jelenlétében , a HNH domén hasította a crRNS-hez hibridizálódott cél DNS-szálat és a RuvC domén hasította a nem-hibridizált DNS-szálat. Az így kapott vágás, amely a PAM-tól körülbelül 3 nm-re történik felfelé a szálon, tompa végű kettős szálú törések kialakulásához vezet . Ezt követően a Cas9 szilárd kapcsolatban marad a vírus DNS tompa végeivel.
A III. Típusú CRISPR / Cas rendszerekben az interferencia-komplex felismeri a kapott RNS-transzkriptumot, amely komplementer a crRNS-távtartó szekvenciájával, és lebontja mind a transzkriptumot, mind a DNS-t, amelyből a transzkriptum keletkezett. Ez a folyamat transzkripciótól függő DNS-interferencia néven ismert. Az interferencia komplexnek három enzimatikus aktivitása van:
- krRNS-irányított endoribonukleáz aktivitás a cél RNS ellen Csm3 (III-A típus) vagy Cmr4 (III-B típus)
- Célzott RNS-stimulált DNáz aktivitás a Cas10 HD doménjén keresztül (Csm1 a III-A és III-D típusokban vagy a Cmr2 a III-B és III-C típusokban)
- Cél-RNS-stimulált cOA (ciklikus oligoadenilát) szintetáz aktivitás a Cas10 "tenyérdoménjén keresztül" (Csm1 a III-A és III-D típusoknál vagy Cmr2 a III-B és III-C típusoknál)
Baktériumok esetén a crmRR-kontrollált Csm (III-A típus) vagy Cmr (III-B típus) komplexek kerülnek az RNS-transzkriptumba, amely beindítja a transzkriptum hasítását a Csm3 vagy Cmr4 alegységek és egyúttal a DNáz a Csm1 vagy Cmr2 aktivitása aktiválódik az ssDNS csatolt lebontására a transzkripciós buborékban. A Csm1 vagy Cmr2 „tenyérdoménje ”, pontosabban a cikláz domén képes az ATP-ből cOA-t termelni, amikor az RNS-transzkriptum meg van kötve. A cOA viszont megköti és aktiválja a Csm6 (III-A típus) vagy a Csx1 (III-B, III-C és III-D típus) ribonukleázokat, hogy növeljék ribonukleáz-aktivitásukat az RNS-átírások lebontása és ezáltal egy további interferencia-mechanizmus kialakítása érdekében .
Hatások
A CRISPR / Cas mechanizmus révén a baktériumok immunitást szerezhetnek bizonyos fágokkal szemben, és továbbadhatják a megszerzett immunitást, mivel egy vírusspecifikus távtartót integrálnak genomjukba, és így továbbadják a replikáció során . Emiatt az a provokatív tézis hangzott el, hogy a CRISPR-Cas rendszer volt az első valóban lamarckista öröklődési mechanizmus.
Alkalmazások
Számos javaslat van a CRISPR biotechnológiai alkalmazására :
- Fágok elleni mesterséges immunizálás megfelelő távtartók hozzáadásával az iparilag fontos baktériumokhoz, pl. B. a tej- és boriparban ,
- Az endogén gének lebontása egy CRISPR régiót tartalmazó plazmiddal történő transzformációval a leállítandó génhez illeszkedő crRNS-sel,
- A multiplex genom szerkesztése lehetővé teszi a különböző célszekvenciák egyidejű mutációját, ami akár két évről néhány hétre is lerövidíti a transzgénikus állatok, például az egerek termelési idejét ,
- Különböző baktériumtörzsek differenciálása a távtartó régiók összehasonlításával ( spoligotipizálás ),
- Génterápia ,
- Fluoreszcens jelölés .
irodalom
- Martin Jinek , Krzysztof Chylinski, Ines Fonfara, Michael Hauer, Jennifer Doudna , Emmanuelle Charpentier : Programozható kettős RNS - irányított DNS-endonukleáz az adaptív baktériumok immunitásában (PDF). In: Science , Vol. 337, No. 6096, 2012. augusztus 17., 816. o., ISSN 0036-8075 . (Angol)
Egyéni bizonyíték
- ↑ Y. Ishino, H. Shinagawa K. Makino M. Amemura A. Nakata A: Az iap gén nukleotidszekvenciája, amely felelős az alkalikus foszfatáz izozim konverzióért Escherichia coliban, és a géntermék azonosítása. Journal of Bacteriology 169: 5429-5433 (1987)
- ^ PM Groenen, AE Bunschoten, D. van Soolingen, JD van Embden: A DNS polimorfizmus természete a Mycobacterium tuberculosis közvetlen ismétlődő klaszterében; törzsdifferenciálás újszerű tipizálási módszerrel történő alkalmazása. Molecular Microbiolology 10, 1993; 1057-1065.
- ↑ FJ Mojica, C. Ferrer, G. Juez, F. Rodriguez-Valera: A rövid tandem ismétlések hosszú szakaszai vannak az Archaea Haloferax mediterranei és a Haloferax volcanii legnagyobb replikációiban, és részt vehetnek a replikon particionálásában. Molecular Microbiology 17, 1995; 85-93.
- ^ FJ Mojica, C. Diez-Villasenor, E. Soria, G. Juez: A rendszeresen elosztott ismétlődések családjának biológiai jelentősége az Archaea, Bacteria és mitochondrium genomjában. Molecular Microbiology 36, 2000; 244-246.
- ↑ a b Sinan Al-Attar, Edze R. Westra, John van der Oos, Stan JJ Brouns: Rendszeresen csoportosított rövid palindrómás ismétlések (CRISPR): a prokariótákban található ötletes vírusellenes védelmi mechanizmus jellemzői. Biológiai Kémia 392, 2011; 277-289. doi: 10.1515 / BC.2011.042 , teljes szöveg
- ↑ R. Jansen, JD Embden, W. Gaastra, LM Schouls: A prokariótákban előforduló DNS ismétlésekkel társult gének azonosítása. Mol Microbiol 43: 1565-1575 (2002)
- ↑ DH Haft, J. Selengut, EF Mongodin, KE Nelson: A prokarióta genomokban 45 CRISPR-asszociált (Cas) fehérjecsaládból és több CRISPR / Cas altípusból álló céh létezik. PLoS Comput. Biol. 1, 2005; e60.
- /1 125/150 Archaea, 1126/2480 Bacteria CRISPRdb , 2013. január 19-én
- ↑ a b T. R. Sampson, DS Weiss: Alternatív szerepek a CRISPR / Cas rendszerek számára a baktériumok patogenezisében. In: PLoS kórokozók. 9. évfolyam, 10. szám, 2013., E1003621, ISSN 1553-7374 . doi: 10.1371 / journal.ppat.1003621 . PMID 24146613 . PMC 3798603 (ingyenes teljes szöveg).
- ↑ a b c Horváth R., Barrangou: CRISPR / Cas, a baktériumok és az archeák immunrendszere. Science 327, 167. o. (2010)
- ↑ LA Marraffini, EJ Sontheimer: CRISPR interferencia: RNS-irányított adaptív immunitás baktériumokban és archeákban. In: Nature Reviews Genetics . 11. évfolyam, 3. szám, 2010. március, 181–190. O., Doi : 10.1038 / nrg2749 , PMID 20125085 , PMC 2928866 (szabad teljes szöveg) (áttekintés).
- ↑ a b c d E. Charpentier, H. Richter, J. van der Oost, MF White: Az RNS-vezetők biogenezis útjai archeealis és bakteriális CRISPR-Cas adaptív immunitásban. In: FEMS mikrobiológiai áttekintések. 39. évfolyam, 3. szám, 2015. május, 428-441. O., Doi : 10.1093 / femsre / fuv023 , PMID 25994611 , PMC 5965381 (ingyenes teljes szöveg) (áttekintés).
- ↑ a b c d e CRISPR Locus. In: Sino Biological. Letöltve: 2020. január 11 .
- ^ FJ Mojica, C. Diez-Villasenor, J. Garcia-Martinez, E. Soria: A rendszeresen elhelyezett prokarióta ismétlések közbeiktatott szekvenciái idegen genetikai elemekből származnak. Journal of Molecular Evolution 60: 174-182 (2005).
- ↑ C. Pourcel, G. Salvignol, G. Vergnaud: CRISPR elemek Yersinia pestis szerezhet új ismétlődés által előnyös felvételét bakteriofág DNS, és a további eszközöket biztosítanak evolúciós vizsgálatok. Microbiology 151: 653-663 (2005).
- ↑ A. Bolotin, B. Quinquis, A. Sorokin, SD Ehrlich: Fürtözött rendszeresen interspaced rövid palindrom ismétlődések (CRISPRs) van távtartók extrakromoszomális eredetű. Microbiology 151: 2551-2561 (2005).
- ↑ Donghyun Ka, Dong Man Jang, Byung Woo Han, Euiyoung Bae: A II-A típusú CRISPR adaptációs modul molekuláris szervezete és kölcsönhatása a Cas9-vel Csn2-n keresztül. In: Nukleinsavak kutatása. 46, 2018, 9805. o., Doi : 10.1093 / nar / gky702 .
- ^ Eugene V. Koonin: CRISPR: a genomtervezés új alapelve, amely az evolúciós biológia fogalmi elmozdulásaihoz kapcsolódik. In: Biológia és filozófia. 2019. évi 34. évfolyam , doi : 10.1007 / s10539-018-9658-7 .
- ↑ Omer S. Alkhnbashi, Shiraz A. Shah, Roger A. Garrett, Sita J. Saunders, Fabrizio Costa, Rolf Ofen: A CRISPR locusok vezetőszekvenciáinak jellemzése. In: Bioinformatika. 32. évfolyam, 17. szám, 2016. szeptember 1., I576 - i585, doi : 10.1093 / bioinformatics / btw454 .
- ^ A b R. Barrangou, C. Fremaux, H. Deveau, M. Richards, P. Boyaval, S. Moineau, DA Romero, Horvath A.: A CRISPR szerzett rezisztenciát nyújt a vírusokkal szemben a prokariótákban. Science 315, 1709-1712 (2007)
- ↑ Jennifer Doudna , Emmanuelle Charpentier .: Az új határ a genom mérnöki CRISPR-Cas9. Science 346 (6213): 1077 (2014), PMID 25430774 , doi: 10.1126 / science.1258096 .
- ↑ CRISPR Spacer beszerzése. In: Sino Biological. Hozzáférés: 2020. január 16 .
- ↑ a b Kirill A. Datsenko, Ksenia Pougach, Anton Tikhonov, Barry L. Wanner, Konstantin Severinov, Ekaterina Semenova: A korábbi fertőzések molekuláris memóriája aktiválja a CRISPR / Cas adaptív bakteriális immunrendszert. In: Nature Communications. 2012. 3. évfolyam , doi : 10.1038 / ncomms1937 .
- ↑ Daan C. Swarts, Cas Mosterd, Mark WJ van Passel, Stan JJ Brouns, Igor Mokrousov: A CRISPR Interference Dirands Strand Specific Spacer Acquisition. In: PLoS ONE. 2012. évfolyam, 7. kötet, E35888. O., Doi : 10.1371 / journal.pone.0035888 .
- ↑ Ido Yosef, Moran G. Goren, Udi Qimron: Az Escherichia coli CRISPR adaptációs folyamatához nélkülözhetetlen fehérjék és DNS-elemek. In: Nukleinsavak kutatása. 40. évfolyam , 2012, 5569. o., Doi : 10.1093 / nar / gks216 .
- ↑ PC Fineran, MJH Gerritzen, M. Suarez-Diez, T. Kunne, J. Boekhorst, Sacha AFT van Hijum, Raymond HJ Staals, Stan JJ Brouns: A degenerált célhelyek gyors primer CRISPR adaptációt közvetítenek. In: A Nemzeti Tudományos Akadémia közleményei. 111. évfolyam, 2014. szám, 16. szám, E1629 - E1638, doi : 10.1073 / pnas.1400071111 .
- B a b Kaylee E. Dillard, Maxwell W. Brown, Nicole V. Johnson, Yibei Xiao, Adam Dolan, Erik Hernandez, Samuel D. Dahlhauser, Yoori Kim, Logan R. Myler, Eric V. Anslyn, Ailong Ke, Ilya J Finkelstein: CRISPR-Cas alapozású beszerzési komplexum összeszerelése és áthelyezése. In: Cell. 175. évfolyam , 2018, 934 o., Doi : 10.1016 / j.cell.2018.09.039 .
- ↑ a b c d S.JJ Brouns, MM Jore, M. Lundgren, ER Westra, RJH Slijkhuis, APL Snijders, MJ Dickman, KS Makarova, EV Koonin, J. van der Oost: Small CRISPR RNSs Guide Antiviral Defense in Prokaryotes . Science 321: 960 (2008)
- ↑ Yibei Xiao, Min Luo, Robert P. Hayes, Jonathan Kim, Sherwin Ng, Fang Ding, Maofu Liao, Ailong Ke: Az irányított R-hurok kialakulásának és az aljzatátadási mechanizmusok szerkezeti alapjai az I. típusú CRISPR-Cas rendszerben. In: Cell. 2017. évi 170. évfolyam , 48. o., Doi : 10.1016 / j.cell.2017.06.012 .
- ↑ Olga Musharova, Evgeny Klimuk, Kirill A. Datsenko, Anastasia Metlitskaya, Maria Logacheva, Ekaterina Semenova, Konstantin Severinov, Ekaterina Savitskaya: A távtartó hosszúságú DNS-köztitermékek a Cas1-hez kapcsolódnak primer CRISPR adaptáción áteső sejtekben. In: Nukleinsavak kutatása. 45. évfolyam , 2017, 3297. o., Doi : 10.1093 / nar / gkx097 .
- ↑ R. Sorek, CM Lawrence, B. Wiedenheft: CRISPR-közvetített adaptív immunrendszere a baktériumok és archaea. In: A biokémia éves áttekintése. 82. évfolyam, 2013, 237-266. O., Doi : 10.1146 / annurev-biochem-072911-172315 , PMID 23495939 (áttekintés).
- ↑ KN Yoganand, R. Sivathanu, S. Nimkar, B. Anand: Asymmetric elhelyezése Cas1-2 komplex és integrációs Host faktor által indukált DNS-hajlító útmutató az egyirányú homing protospacer a CRISPR-Cas típusú IE rendszer. In: Nucleic Acids Research. 45. évfolyam, 1. szám, 2017. január, 367-381. Oldal, doi : 10.1093 / nar / gkw1151 , PMID 27899566 , PMC 5224486 (ingyenes teljes szöveg).
- ^ Y. Xiao, S. Ng, KH Nam, A. Ke: Hogyan alakítja ki a II típusú CRISPR-Cas az immunitást a Cas1-Cas2 által közvetített távtartó integráció révén. In: Természet . 550. évfolyam, 7674. szám, 2017. 10., 137–141. Oldal, doi : 10.1038 / nature24020 , PMID 28869593 , PMC 5832332 (ingyenes teljes szöveg).
- ↑ J. McGinn, LA Marraffini: A CRISPR-Cas rendszerek optimalizálják immunválaszukat azáltal, hogy meghatározzák a távtartó integráció helyét. In: Molekuláris sejt. 64. évfolyam , 3. szám, 2016. november , 616–623. Oldal, doi : 10.1016 / j.molcel.2016.08.038 , PMID 27618488 , PMC 5096952 (ingyenes teljes szöveg).
- ↑ JK Nuñez, AS Lee, A. Engelman, JA Doudna: Integráz által közvetített távtartó felvétel a CRISPR-Cas adaptív immunitás alatt. In: Természet . Kötet 519, szám 7542, március 2015, pp. 193-198, doi : 10.1038 / nature14237 , PMID 25.707.795 , PMC 4359072 (ingyenes teljes szöveg).
- B a b Jenny G Kim, Sandra Garrett, Yunzhou Wei, Brenton R. Graveley, Michael P. Terns: CRISPR DNS elemek, amelyek helyspecifikus távtartó integrációt és megfelelő ismétlési hosszúságot vezérelnek egy II. Típusú CRISPR-Cas rendszerrel. In: Nucleic Acids Research . 47. évfolyam , 2019., 8632. o., Doi : 10.1093 / nar / gkz677 .
- ^ MG Goren, S. Doron, R. Globus, G. Amitai, R. Sorek, U. Qimron: Ismételje meg a méret meghatározását két molekuláris vonalzóval az IE CRISPR típusú tömbben. In: Cella jelentések. 16. évfolyam, 11. szám, 2016. szeptember, 2811-2818. O., Doi : 10.1016 / j.celrep.2016.08.043 , PMID 27626652 , PMC 5039180 (ingyenes teljes szöveg).
- ^ SH Sternberg, H. Richter, E. Charpentier, U. Qimron: Adaptáció a CRISPR-Cas rendszerekben. In: Molekuláris sejt. 61. évfolyam, 6. szám, 2016. március, 797–808. Oldal, doi : 10.1016 / j.molcel.2016.01.030 , PMID 26949040 (áttekintés).
- B a b J. Carte, R. Wang, H. Li, RM Terns , MP Terns : A Cas6 egy endoribonukleáz, amely vezető RNS-eket generál a betolakodók védelmére prokariótákban. In: Gének és fejlődés. 22. évfolyam, 24. szám, 2008. december, 3489-3496. O., Doi : 10.1101 / gad.1742908 , PMID 19141480 , PMC 2607076 (ingyenes teljes szöveg).
- ↑ a b c MM Jore, M. Lundgren, E. van Duijn, JB Bultema, ER Westra, SP Waghmare, B. Wiedenheft, U. Pul, R. Wurm, R. Wagner, Beijer úr, A. Barendregt, K. Zhou, AP Snijders, MJ Dickman, JA Doudna, EJ Boekema, AJ Heck, J. van der Oost, SJ Brouns: Strukturális alap a CRISPR RNS-vezérelt DNS-felismeréséhez Cascade. In: Természet szerkezeti és molekuláris biológia. 18. évfolyam , 5. szám, 2011. május , 529–536 . Oldal, doi : 10.1038 / nsmb.2019 , PMID 21460843 .
- ↑ E. Deltcheva, K. Chylinski, CM Sharma, K. Gonzales, Y. Chao, ZA Pirzada, MR Eckert, J. Vogel, E. Charpentier: CRISPR RNS érése transzkódolt kis RNS és gazda faktor RNáz III. In: Természet . 471. évfolyam, 7340. szám, 2011. március, 602-607. O., Doi : 10.1038 / nature09886 , PMID 21455174 , PMC 3070239 (ingyenes teljes szöveg).
- ↑ Y. Zhang, N. Heidrich, BJ Ampattu, CW Gunderson, HS Seifert, C. Schoen, J. Vogel, EJ Sontheimer: A feldolgozástól független CRISPR RNS-ek korlátozzák a Neisseria meningitidis természetes átalakulását. In: Molekuláris sejt. 50. évfolyam, 4. szám, 2013. május, 488-503. O., Doi : 10.1016 / j.molcel.2013.05.001 , PMID 23706818 , PMC 3694421 (ingyenes teljes szöveg).
- ↑ M. Jinek, K. Chylinski, I. Fonfara, M. Hauer, JA Doudna, E. Charpentier: Programozható kettős RNS-vezérelt DNS endonukleáz adaptív bakteriális immunitásban. In: Tudomány . 337. évfolyam, 6096. szám, 2012. augusztus, 816–821. O., Doi : 10.1126 / science.1225829 , PMID 22745249 , PMC 6286148 (ingyenes teljes szöveg).
- Cart J. Carte, NT Pfister, MM Compton, RM Terns, MP Terns: A CRISPR RNS megkötése és hasítása Cas6 által. In: RNS. 16. évfolyam, 11. szám, 2010. november, 2181-2188. O., Doi : 10.1261 / rna.2230110 , PMID 20884784 , PMC 2957057 (ingyenes teljes szöveg).
- ↑ KS Makarova, DH Haft, R. Barrangou, SJ Brouns, E. Charpentier, Horváth P., S. Moineau, FJ Mojica, YI Wolf, AF Yakunin, J. van der Oost, EV Koonin: A CRISPR alakulása és osztályozása -Cas rendszerek. In: Természet vélemények. Mikrobiológia. 9. évfolyam, 6. szám, 2011. 06., 467–477. Oldal, doi : 10.1038 / nrmicro2577 , PMID 21552286 , PMC 3380444 (szabad teljes szöveg) (áttekintés).
- ↑ a b A. Plagens, H. Richter, E. Charpentier, L. Randau: DNS- és RNS-interferencia mechanizmusok CRISPR-Cas megfigyelési komplexekkel. In: FEMS mikrobiológiai áttekintések. 39. évfolyam, 3. szám, 2015. május, 442–463. O., Doi : 10.1093 / femsre / fuv019 , PMID 25934119 , PMC 5965380 (ingyenes teljes szöveg) (áttekintés).
- ↑ RN Jackson, SM Golden, PB van Erp, J. Carter, ER Westra, SJ Brouns, J. van der Oost, TC Terwilliger, RJ Read, B. Wiedenheft: Strukturális biológia. Az Escherichia coli-ból származó CRISPR RNS-vezérelt megfigyelési komplex kristályszerkezete. In: Tudomány . 345. évfolyam , 6203. szám, 2014. szeptember , 1473–1479 . O., Doi : 10.1126 / science.1256328 , PMID 25103409 , PMC 4188430 (ingyenes teljes szöveg).
- ↑ B. Wiedenheft, GC Lander, K. Zhou, MM Jore, SJ Brouns, J. van der Oost, JA Doudna, E. Nogales: Az RNS-vezérelt megfigyelési komplex struktúrája egy bakteriális immunrendszerből. In: Nature . 477. évfolyam, 7365. szám, 2011. szeptember, 486–489. Oldal, doi : 10.1038 / nature10402 , PMID 21938068 , PMC 4165517 (ingyenes teljes szöveg).
- ↑ DG Sashital, B. Wieden könyv, IGEN Doudna: Az idegen DNS mechanizmusa baktériumok szelekciós adaptív immunrendszerévé. In: Molecular cell. 46. évfolyam, 5. szám, 2012. június, 606–615. O., Doi : 10.1016 / j.molcel.2012.03.020 , PMID 22521690 , PMC 3397241 (ingyenes teljes szöveg).
- ^ ER Westra, PB van Erp, T. Künne, SP Wong, RH Staals, CL Seegers, S. Bollen, MM Jore, E. Semenova, K. Severinov, WM de Vos, RT Dame, R. de Vries, SJ Brouns , J. van der Oost: A CRISPR immunitás a negatívan szupertekercselt betolakodó DNS Cascade és Cas3 általi egymás utáni megkötésén és lebontásán alapul. In: Molekuláris sejt. 46. évfolyam, 5. szám, 2012. június, 595–605. O., Doi : 10.1016 / j.molcel.2012.03.018 , PMID 22521689 , PMC 3372689 (ingyenes teljes szöveg).
- ↑ a b B. Gong, M. Shin, J. V., CH Jung, EL Bolt, J. van der Oost, JS Kim: Molecular betekintést DNS interferenciát CRISPR-asszociált nukleáz-helikáz Cas3. In: A Nemzeti Tudományos Akadémia közleményei . 111. évfolyam, 46. szám, 2014. november, 16359-16364, doi : 10.1073 / pnas.1410806111 , PMID 25368186 , PMC 4246338 (ingyenes teljes szöveg).
- ^ A b Y. Huo, KH Nam, F. Ding, H. Lee, L. Wu, Y. Xiao, MD Farchione, S. Zhou, K. Rajashankar, I. Kurinov, R. Zhang, A. Ke: Szerkezetek A CRISPR Cas3 of Mechanical betekintést nyújt a Cascade-aktivált DNS-letekerésbe és lebontásba. In: Természet szerkezeti és molekuláris biológia. 21. évfolyam, 9. szám, 2014. szeptember, 771-777. Oldal, doi : 10.1038 / nsmb.2875 , PMID 25132177 , PMC 4156918 (ingyenes teljes szöveg).
- B a b S. Mulepati, S. Bailey: Az Escherichia coli RNS-vezérelt immunrendszer in vitro rekonstrukciója a DNS-célirány egyirányú, ATP-függő lebomlását tárja fel. In: Journal of Biological Chemistry . 288. évfolyam, 31. szám, 2013. augusztus, 22184-22192. O., Doi : 10.1074 / jbc.M113.472233 , PMID 23760266 , PMC 3829311 (ingyenes teljes szöveg).
- ↑ a b T. Sinkunas, G. Gasiunas, C. Fremaux, R. Barrangou, P. Horváth, V. Siksnys: Cas3 egy egyszálú DNS-nukleáz és az ATP-függő helikázt a CRISPR / Cas immunrendszert. In: Az EMBO folyóirat . 30. évfolyam, 7. szám, 2011. április, 1335-1342. O., Doi : 10.1038 / emboj.2011.41 , PMID 21343909 , PMC 3094125 (ingyenes teljes szöveg).
- ↑ M. Jinek, F. Jiang, DW Taylor, SH Sternberg, E. Kaya, E. Ma, C. Anders, M. Hauer, K. Zhou, S. Lin, M. Kaplan, AT Iavarone, E. Charpentier, E. Nogales, JA Doudna: A Cas9 endonukleázok szerkezete RNS-közvetített konformációs aktivációt tár fel. In: Tudomány . 343. évfolyam, 6176. szám, 2014. március, 1247997 o., Doi : 10.1126 / science.1247997 , PMID 24505130 , PMC 4184034 (ingyenes teljes szöveg).
- ^ A b C. Anders, O. Niewoehner, A. Duerst, M. Jinek: A PAM-függő cél DNS Cas9 endonukleáz általi felismerésének strukturális alapjai. In: Természet . 513. kötet, 7519. szám, 2014. szeptember, 569–573. O., Doi : 10.1038 / nature13579 , PMID 25079318 , PMC 4176945 (ingyenes teljes szöveg).
- ↑ a b H. Nishimasu, FA Ran, PD Hsu, S. Konermann, SI Shehata, N. Dohmae, R. Ishitani, F. Zhang, O. Nureki: Cas9 kristályszerkezete a vezető RNS-sel és a cél DNS-sel komplexben. In: Cell . 156. évfolyam, 5. szám, 2014. február, 935–949. O., Doi : 10.1016 / j.cell.2014.02.001 , PMID 24529477 , PMC 4139937 (ingyenes teljes szöveg).
- ↑ a b Elitsa Y. Dimova, Thomas Kietzmann: Genomváltozások - CRISPR / Cas9 mint választott módszer vagy agónia? In: BIOspectrum. 2018. 24. 24., 702. o., Doi : 10.1007 / s12268-018-0977-7 .
- ↑ a b S. H. Sternberg, S. Redding, M. Jinek, EC Greene, JA Doudna: DNS-kihallgatás a CRISPR RNS-vezérelt Cas9 endonukleázzal. In: Természet . 507. évfolyam , 7490. szám, 2014. március, 62–67. Oldal, doi : 10.1038 / nature13011 , PMID 24476820 , PMC 4106473 (ingyenes teljes szöveg).
- ^ RA Forties, R. Bundschuh, MG Poirier: A lokálisan megolvadt DNS rugalmassága. In: Nukleinsavak kutatása. 37. évfolyam, 14. szám, 2009. augusztus, 4580-4586. Oldal, doi : 10.1093 / nar / gkp442 , PMID 19487242 , PMC 2724272 (ingyenes teljes szöveg).
- ↑ R. Sapranauskas, G. Gasiunas, C. Fremaux, R. Barrangou, P. Horvath, V. Siksnys: A Streptococcus thermophilus CRISPR / Cas rendszer immunitást biztosít Escherichia coliban. In: Nukleinsavak kutatása. 39. évfolyam, 21. szám, 2011. november, 9275–9282. Oldal, doi : 10.1093 / nar / gkr606 , PMID 21813460 , PMC 3241640 (ingyenes teljes szöveg).
- ^ S. Silas, P. Lucas-Elio, SA Jackson, A. Aroca-Crevillén, LL Hansen, PC Fineran, AZ Fire, A. Sánchez-Amat: A III. Típusú CRISPR-Cas rendszerek képesek redundanciát nyújtani a vírusos típusú menekülés ellensúlyozására I rendszerek. In: eLife. 6. kötet, 2017. 08., S., doi : 10.7554 / eLife.27601 , PMID 28826484 , PMC 5576922 (ingyenes teljes szöveg).
- ↑ a b típusú CRISPR-Cas rendszerek. In: Sino Biological. Letöltve: 2019. február 14 .
- ↑ Rafael Molina, Stefano Stella, Mingxia Feng, Nicholas Sofos, Vykintas Jauniskis, Irina Pozdnyakova, Blanca López-Méndez, Qunxin She, Guillermo Montoya: A Csx1-cOA4 komplex felépítése feltárja az RNS bomlás alapját a III-B típusú CRISPR- . In: Nature Communications. 2019. 10., doi : 10.1038 / s41467-019-12244-z .
- ↑ EV Kooni, YI Wolf: Az evolúció darwini vagy / és Lamarck-i? . Biology Direct 4:42 (2009)
- ^ R. Sorek, V. Kunin, P. Hugenholtz: CRISPR - széles körben elterjedt rendszer, amely megszerzett rezisztenciát biztosít a baktériumok és az archeák fágjaival szemben . Nat Rev Microbiol 6: 181 (2007).
- ↑ H. Wang, H. Yang, CS Shivalila, MM Dawlaty, AW Cheng, F. Zhang, R. Jaenisch A több génben mutációt hordozó egerek egylépcsős generációja CRISPR / Cas-Mediated Genome Engineering , Cell, 153. kötet, 4. szám, 2013. május 9., 910–918. Oldal.